Variantes Génétiques du Virus (Max Lafontan)
Variantes génétiques du SARS-CoV-2 – Que signifient-elles ?
(mutation, variante(variant) et souche(clade))
La « perfide Albion », dopée au Brexit, est sur la sellette et nage dans une sérieuse confusion face à l’épidémie de la COVID-19. La voilà confrontée à un nouveau variant du virus SARS-CoV-2 : ses hôpitaux débordent et des Anglais meurent par déficit de soins et malgré une campagne intense de vaccinations qui ne donnera pas de résultats vraiment tangibles avant des mois.
La peur se répand dans de nombreux pays, tandis que surgissent de nouveaux variants (on parle de clades phylogénétiques variants) en Angleterre, en Afrique du Sud, au Brésil, au Japon… L’arbre phylogénétique du virus se ramifie de plus en plus (voir le schéma ci-dessous).
- Mutations, variantes et propagation
Depuis le début de la pandémie de la COVID-19 où en sommes-nous dans le monde ? Le CSSE (Center for Systems Science and Engineering) de la Johns Hopkins University (COVID-19 Map – Johns Hopkins Coronavirus Resource Center (jhu.edu) affiche quotidiennement un suivi régulier dans 191 pays et régions. Le 16/1/2021, nous en sommes à 2 017 380 morts dans le monde depuis la signalisation des premiers cas de l’apparition de la maladie par le bureau de l’OMS en Chine fin décembre 2019.
Autre adresse très utile pour les chercheurs, les praticiens et les curieux anglophones, le site internet Covid-nma.com, (COVID-NMA : Covid-19 open living evidence synthesis to inform decision – Living mapping and living systematic review of Covid-19 trials) animé par le Centre d’épidémiologie clinique de l’Hôtel-Dieu (APHP Paris), l’Inserm, le CNRS, Cochrane France, Germany, Chile…et de nombreux autres partenaires internationaux. Il est actualisé chaque semaine pour répertorier les protocoles en cours et quotidiennement pour les publications. Des centaines de synthèses et de publications en accès direct ! (à titre d’exemple, au 11 janvier il y a 2 409 essais randomisés sur la plateforme de la WHO (actualisé chaque semaine) et 166 articles et prépublications (actualisé chaque jour). Ne pas s’y noyer ! Garder la tête froide ! Tenir le cap sans se démoraliser ! Fluctuat nec mergitur !
Du Royaume-Uni, d’Afrique du Sud et maintenant d’Amazonie brésilienne : de nouvelles variantes du virus SARS-CoV-2 responsable de la Covid-19 se propagent dans le monde. Elles vont être l’objet, le jeudi 14 janvier 2021, d’une réunion du comité d’urgence de l’Organisation Mondiale de la Santé (OMS).
La résurgence du virus frappe également la Chine, qui avait largement maîtrisé l’épidémie mais qui a recensé ce jeudi son premier décès dû au coronavirus depuis mai dernier. Il est difficile de décrypter les informations venant de Chine
Face à l’apparition de variants plus contagieux du coronavirus, le comité d’urgence de l’Organisation mondiale de la santé (OMS) devrait émettre très rapidement des recommandations. Il faut faire vite car les variants plus contagieux sont susceptibles de provoquer une nouvelle explosion de la pandémie.
Précisons certains termes avant d’examiner ces diverses péripéties virales. Les mutations apparaissent car elles sont un sous-produit naturel de la réplication virale. Les virus à ARN sont généralement connus pour avoir un taux de mutation plus élevé que les virus à ADN. Les enzymes impliquées dans la réplication du matériel génétique de ces virus sont « des réplicases ». Elles sont responsables de la variabilité des virus car leur précision de copie est plus ou moins précise. Les coronavirus (qui sont des virus à ARN comme SARS-CoV-2), font cependant moins de mutations que la plupart des virus à ARN connus car ils codent pour une « enzyme correctrice » qui est capable de corriger certaines des erreurs introduites par « les réplicases » lors de la réplication, mais sans les empêcher totalement !
Malgré ce dispositif protecteur (correcteur), il va fait apparaître des erreurs. Si elles lui sont défavorables le virus variant va décliner. Soit, elles lui sont favorables, elles vont accroître sa contagiosité et/ou sa dangerosité. Un virus est simplement confronté à une alternative simple : survivre ; et optimiser ses conditions de survie. Nous sommes face à un mécanisme darwinien incontournable et implacable : les mutants les plus efficaces finissent toujours par prendre le dessus.
Dans la plupart des cas, le sort d’une mutation nouvellement apparue est déterminé par la sélection naturelle.On peut comprendre aisément que les mutations bénéfiques, celles qui vont conférer un avantage concurrentiel vis-à-vis de la réplication virale, de la transmission ou l’évitement de l’immunité des hôtes sont susceptibles d’augmenter en fréquence. Par contre les mutations qui amputent les capacités virales auront tendance à être éliminées de la population de virus en circulation.
Cependant, des mutations peuvent également augmenter et diminuer en fréquence en raison d’événements totalement fortuits. Par exemple, un « effet fondateur » se produit lorsqu’un nombre limité de virus individuels établit une nouvelle population pendant la transmission. Les mutations présentes dans les génomes de ces ancêtres viraux dominent la population indépendamment de leurs effets sur l’aptitude virale. Cette interaction entre les processus de sélection naturelle et les événements fortuits vont façonner l’évolution du virus chez l’hôte, et donc l’histoire de la pandémie au sein de diverses communautés de proximité qu’entre les pays. Il faut remarquer un point important : plus le nombre de personnes touchées s’accroît, plus le risque de mutations augmente. Notre organisme essaie de contrôler le virus en développant une réponse immunitaire adaptée à la forme virale qui l’envahit. Mais de son côté, le virus va tenter d’échapper à cette réponse immunitaire en mutant.
Bien que les termes mutation, variante (ou variant) et souche soient souvent utilisés de manière interchangeable pour décrire le contexte épidémiologie du SARS-CoV-2, les distinctions dans les termes utilisés sontimportantes. Une mutation fait référence au changement qui apparaît dans le gène viral et dans la séquence d’acides aminés de la protéine codée par le gène viral. Elle peut être due à une substitution d’un seul nucléotide de la séquence du gène jusqu’à une délétion d’une partie du gène qui se solde par la perte de quelques acides aminés (mentionnée par del —) de la protéine. Des mutations des triplets de nucléotides vont déboucher sur des substitutions d’acides aminés : par exemple D614G est une substitution d’un acide aspartique (D) en glycine (G) en position 614 de la glycoprotéine de pointe. Certaines mutations qui ne concernent qu’un seul nucléotide du gène (ARNm de SARS-CoV-2) peuvent être sans incidence notable. Les curieux peuvent consulter le site internet du consortium GISAID (https://www.gisaid.org/), qui a été mis en place dès 2008 pour partager les données virales lors de l’épidémie de grippe aviaire (H5N1) qui héberge à ce jour la plus grande collection de génomes de SARS-CoV-2 et des répertoires de mutations/délétion (plus de 370 000 documents soumis par les chercheurs virologues).
Les génomes qui diffèrent dans l’ordre des séquences sont souvent appelés variantes (ou variants). Ce terme est un peu moins précis car deux variantes peuvent différer d’une mutation ou de plusieurs mutations. Strictement parlant, une variante est une souche ou clade phylogénétique quand elle a un phénotype différent (par exemple, une différence d’antigénicité, de transmissibilité ou virulence, de résistance au système immunitaire de l’hôte qu’elle va envahir). Certains parlent de « clone ». Certains virologues préfèrent parler de « mutant » plutôt que de « variant » même si le mot peut faire peur à certains.
Avant de sombrer dans la panique ou des délires médiatiques et de se laisser submerger par les terminologies d’experts, retenons qu’une évaluation sérieuse et rapide de tout nouveau variant du SARS-CoV-2 est nécessaire. Elle devrait inclure une évaluation rigoureuse des questions suivantes :
(1) Est-ce que le variant est susceptible de devenir prédominant grâce à la sélection naturelle ou à des événements fortuits ?
(2) Si les preuves suggèrent une sélection naturelle, quelle (s) mutation (s) ont été sélectionnées ?
(3) Quel est l’avantage adaptatif qui est procuré par ces mutations ?
(4) Quel effet réel ces mutations ont-elles sur la transmissibilité et la propagation, l’antigénicité ou le niveau de virulence du virus ?
(5) Ces mutations vont-elles déboucher sur l’inefficacité des anticorps neutralisants produits par le système immunitaire d’un individu vacciné.
- La mutation D614G dans la glycoprotéine de pointe de SARS-CoV-2 (Spike D614G).
La mutation D614G dans la glycoprotéine de pointe de SARS-CoV-2 a été détectée pour la première fois à un niveau significatif dès le début mars 2020 et s’est propagée pour atteindre une domination mondiale sur le mois suivant. La mutation semblait initialement survenir et balayer indépendamment et simultanément plusieurs régions géographiques. Cette apparente convergente suggérait une sélection naturelle et un avantage adaptatif de la forme D614G.
Les efforts de séquençage et d’identification de la mutation D614G dans les virus ont été effectués dans plusieurs provinces chinoises dès la fin janvier. Cette observation a débouché sur l’hypothèse que la possibilité d’une dispersion mondiale de cette mutation aurait pu résulter d’événements fondateurs fortuits « founder events by chance », dans lesquels les virus hébergeant 614G vont être à l’origine de la majorité des premières transmissions.
Récemment dans la traque aux changements qui peuvent apparaître dans le génome de SARS-CoV-2. Korber B. et ses collaborateurs ont découvert et publié dans la revue Cell, des données sur la mutation D614G qui est importante car elle affecte le niveau d’infectiosité des virus qui la véhiculent. En effet, un variant du SARS-CoV-2 qui porte sur le changement d’un acide aminé de la protéine de pointe (protéine S). D614G est devenu la forme la plus répandue dans la pandémie mondiale comme indiqué dans le diagramme ci-dessous. Le changement s’est produit même dans les épidémies locales où la forme D614 d’origine était bien établie avant l’introduction de la variante G614. L’augmentation très rapide du variant G614 au niveau régional puis mondial pourrait indiquer un avantage adaptatif de cette forme virale. Il faut noter que le variant G614 est associé à des seuils inférieurs de cycles (CT) de RT-PCR (plus de virus). C’est un résultat qui suggère que les voies respiratoires supérieures ont une charge virale augmentée (sans qu’il soit constaté un accroissement de la gravité de la COVID-19). Ces travaux mettent en lumière que des changements d’apparence mineure dans la structure de la glycoprotéine S peuvent être importants et aider pour une meilleure appréhension mécaniste de la structure et du comportement du virus. Ils incitent à soutenir la surveillance continue des mutations de la protéine Spike pour aider au développement des interventions immunologiques futures (C’est un point important pour les nombreux vaccins qui ciblent cette protéine, en particulier).
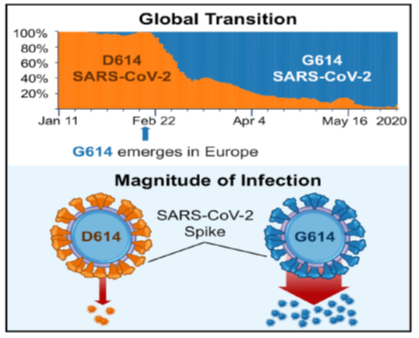
Diagramme synthétique sur l’émergence du variant G614 et de la transition globale en Europe (Extrait de l’article publié dans Cell.
La possibilité d’une telle hypothèse nulle a conduit beaucoup de chercheurs de la communauté impliquée dans des travaux sur l’évolution du virus à douter de ce que la mutation D614G soit adaptative, malgré les données in vitromontrant ses incidences sur la liaison aux récepteurs du virus l’ACE2 (enzyme de conversion de l’angiotensine 2). Une étude génétique et une analyse phylodynamique d’envergure sur une population récente de plus de 25 000 séquences au Royaume-Uni ont révélé que les virus portant 614G ont semblé se propager plus rapidement et semer plus de clusters phylogénétiques que les virus avec 614D. Cet effet est assez modeste et les différents modèles n’ont pas toujours de signification statistique. Plus récemment, un travail complémentaire sur des modèles animaux a indiqué que les virus 614G se transmettent plus efficacement. Cette mutation a eu une très large diffusion planétaire (voir diagramme et article), c’était déjà un virus différent des premiers identifiés en Chine.
- Protéine S, mutation N453Y et visons.
Des épidémies de SARS-CoV-2 ont commencé à apparaître dans les fermes de visons aux Pays-Bas et au Danemark à la fin du printemps et au début de l’été 2020. L’enquête génomique et épidémiologique sur une épidémie précoce dans les Pays-Bas a démontré une transmission de l’homme au vison (Mustela lutreola), du vison à ses propres congénères et du vison à l’homme. Au début novembre 2020 les autorités danoises ont signalé 214 cas de la COVID-19 humaine en relation avec des fermes d’élevage de visons. De nombreuses séquences de SARS-CoV-2 dans des foyers infectieux aux Pays-Bas et au Danemark, portent la mutation Y453F : substitution de la (tyrosine (Y) remplacée par la phenyl alanine (F) en position 453). Elle est localisée dans le domaine de liaison de la protéine S au récepteur. Cette mutation pourrait induire un accroissement des capacités de liaison du virus au récepteur chez le vison (i.e. affinité de liaison accrue à l’ACE2 du vison). Onze personnes de l’épidémie danoise avaient une variante appelée cluster 5, qui avait 3 mutations supplémentaires dans la protéine S (délétion 69_70, mutation I692V [Iso692Val] et M1229I [Met1229Iso]). Une étude initiale de 9 échantillons de sérum provenant de convalescents humains (portant des anticorps) a suggéré une réduction modeste mais significative de l’activité de neutralisation des anticorps de ce sérum contre les virus du cluster 5 qui serait susceptible d’induire un accroissement d’affinité pour L’ACE2 du vison[1].
- Lignée B.1.1.7 et N501Y [Asn501Tyr] anglaise.
La lignée B.1.1.7 (également appelée 501Y.V1) : C’est un cluster phylogénétique qui se propage rapidement dans le sud-est de l’Angleterre. Noter qu’il s’était déjà accumulé dix-sept mutations définissant la lignée avant sa détection au début septembre, ce qui suggère une évolution antérieure significative (éventuellement dans un hôte infecté de manière chronique). On remarquera les efforts notables, disons même exceptionnels, du Royaume-Uni dans les activités de séquençage du génome de variants qui permettent l’identification rapide de mutants. Au 28 décembre 2020, cette variante représentait environ 28 % des cas d’infection par le SARS-CoV-2 en Angleterre et les modèles génétiques d’évolution de cette mutation dans la population suggèrent qu’elle se propage 56 % plus rapidement que les autres lignées virales. Contrairement au D614G, qui aurait vraisemblablement pu bénéficier d’un événement fortuit, la lignée B.1.1.7 s’est étendue lorsque les cas de SARS-CoV-2 étaient répandus. Elle a apparemment atteint un niveau de domination en devançant une population existante des autres variants viraux circulants. … Spécificité de ce virus, on ne retrouve pas les variations saisonnières de la grippe.
Ces données suggèrent fortement l’existence d’une sélection naturelle d’un virus plus transmissible au niveau de la population. L’impact de ce variant sur le nombre de morts, affirmé par Boris Johnson, reste contesté[2]. Alors que les interventions de santé publique en cours comme l’usage des masques, la distanciation physique et les limitations sur les grands rassemblements sont déjà en cours et devraient rester efficaces, le contrôle d’une variante plus transmissiblenécessitera probablement une application encore plus rigoureuse et généralisée dans l’adoption de ces mesures.
Le variant Britannique (B.1.1.7 ou VUI-202012/01) se distingue par un nombre anormalement élevé de mutations dont plusieurs intéressent la protéine S. En fait, huit des mutations de la lignée B.1.1.7 se situent dans la glycoprotéine de pointe (protéine S), y compris N501Y [Asn501Tyr] localisée dans le domaine de liaison au récepteur. La délétion 69_70, et P681H [Pro681His] se situent dans le site de clivage de la furine (référence à un site particulier dans la structure de la protéine). Toutes ces mutations pourraient influencer de manière plausible la liaison au récepteur ACE2 et donc les capacités de charge des cellules et de réplication virale.
Ces données suggèrent fortement l’existence d’une sélection naturelle d’un virus plus transmissible au niveau de la population. Alors que les interventions de santé publique comme les masques, la distanciation physique et les limitations sur les grands rassemblements devraient rester efficaces. Le contrôle d’une variante plus transmissiblenécessitera probablement une application plus rigoureuse et généralisée dans l’adoption de ces mesures.
La diffusion du nouveau variant de SARS-CoV-2 au Royaume Uni :
A, arbre phylogénétique[3] montrant la relation de la lignée B.1.1.7 (20B / 501Y.V1, branche orange et points) avec d’autres lignées circulantes. La longue longueur de branche de cette lignée reflète le fait qu’elle a accumulé un nombre important de mutations avant d’être découverte. Définition « savante » du terme clade en note de bas de page[4].
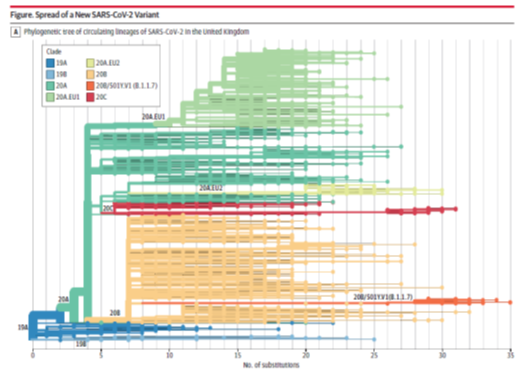
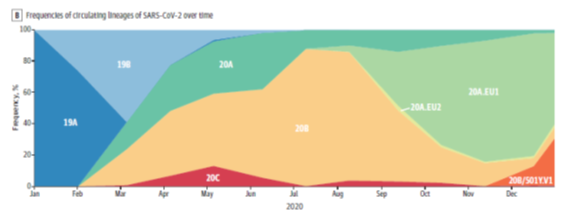
B, Fréquences des lignées circulantes de SARS-CoV-2 dans le temps. Les lignées sont colorées comme dans l’arbre ci-dessus, la lignée B.1.1.7 (20B / 501Y.V1) est représentée en orange
(JAMA, January 6, 2021)
Le variant Britannique (B.1.1.7 ou VUI-202012/01) se distingue par un nombre anormalement élevé de mutations dont plusieurs intéressent la protéine S. En fait, huit des mutations de la lignée B.1.1.7 se situent dans la glycoprotéine de pointe (protéine S), y compris N501Y [Asn501Tyr] localisée dans le domaine de liaison au récepteur. La délétion 69_70, et P681H [Pro681His] se situent dans le site de clivage de la furine (référence à un site particulier dans la structure de la protéine). Toutes ces mutations pourraient influencer de manière plausible la liaison au récepteur ACE2 et donc les capacités de charge des cellules et de réplication virale.
Les variants devraient avoir une affinité plus élevée pour l’ACE2 humaine. Notons, en conclusion de ce paragraphe que le virus britannique n’est ni plus dangereux, ni plus pathogène, mais qu’il est plus transmissible et qu’il risque de créer un engorgement des hôpitaux qui peut déboucher sur des carences de soins et des morts. Le Pr. Bruno Lina, qui coordonne au niveau national la cartographie de la circulation des virus remarque que le « mutant »britannique, retrouvé aujourd’hui dans 1,3 % à 1,4 % des tests positifs au Covid-19 en France, devrait dominer dans deux à trois mois : « C’est une bascule inéluctable » déclare-t-il. Comme la France ne brille pas (elle est même en queue de peloton), par ses capacités de séquençage des génomes de formes émergentes de SARS-CoV-2, il se pourrait que nous soyons très en dessous de la réalité de la présence de ce variant dans la population.
Un autre variant différent (E484K), portant également avec une mutation N501Y, se propage rapidement en Afrique du Sud. Les effets de ces mutations sur l’antigénicité ne sont pas clairs pour le moment.
- Un autre variant qui sévit en Afrique du Sud fait parler de lui…
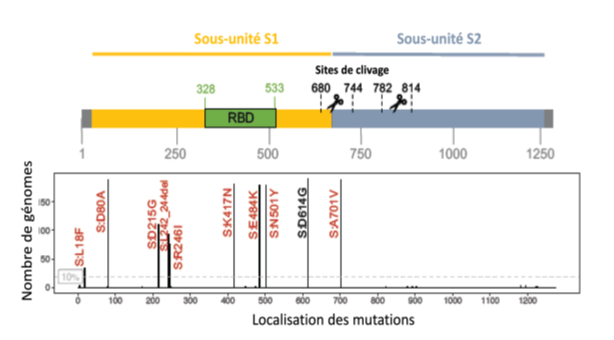
Ce variant de SARS-CoV-2 a lui aussi un nombre élevé de mutations. Le variant (nommé par son clade phylogénétique 501Y.V2) (l’anglais étant appelé 501Y.V1) est devenu très rapidement majoritaire en Afrique de Sud et serait lui aussi, plus transmissible. Il est caractérisé par 8 mutations sur la protéine S. Trois d’entre-elles pourraient être plus inquiétantes car elles se situent sur le site de fixation du Receptor Binding Domain [RBD] de la protéine S à son récepteur ACE2. Les analyses structurales de conformation de la protéine le localisent de la position 328 à 533 des acides aminés). Il faut remarquer que ce RBD se trouve être aussi la cible des vaccins à ARNm actuellement administrés (Pfizer/BioNTech et Moderna). Rappelons les mutations identifiées :
• K417N : porte sur la substitution de l’acide aminé Lysine (K / Lys) par l’Asparagine (N / ASN) en position 417
• E484K : substitution de l’acide aminé Acide Glutamique (E /Glu) par la Lysine (K / Lys)
en position 484
• Mutation N501Y : substitution de l’acide aminé Asparagine (N / Asn) par la Tyrosine (Y ou Tyr) en position 501 (remarquons que cette mutation est aussi présente dans le variant anglais cité ci-dessus)
Si un variant a une ou plusieurs mutations de la protéine S qui augmentent la transmissibilité du virus, il pourrait rapidement surpasser et remplacer d’autres variantes virales. En plus de la mutation déjà identifiée dans le variant anglais (N501Y), les modifications structurales de la protéine dues à la mutation E494K pourraient permettre au virus d’échapper à la réponse immunitaire selon certains auteurs. La question reste débattue et nécessite des travaux plus approfondis qui ne sauraient tarder.
Il est certain que ces mutations posent le problème d’une possibilité de sélection d’un variant privilégié qui pourrait devenir un véritable danger si les modifications structurales induites par ces mutations des acides aminés de la protéine S lui font acquérir une résistance (i.e., non-reconnaissance) aux anticorps produits chez les patients vaccinés par les vaccins actuels.
Plus on laissera le temps au virus de se répandre et de se reproduire dans les cellules des infectés, moins on créera de protections immunitaires (anticorps et cellules mémoire) par des interventions vaccinales, plus le virus aura « le temps » de faire de nouvelles mutations et nous verrons apparaître des clones ou clades phylogénétiques nouveaux.
Schéma : Protéine S de la souche mutée Sud-Africaine représentée par la sous-unité S1 (jaune) et lasous-unité S2 (grisé). Les 8 mutations de substitution d’acides aminés sont mentionnées en rouge ; troisd’entre-elles (K417N, E484K et N501Y) intéressent la région de fixation de la protéine sur le récepteur ACE2 (RBD en vert). Les nombres représentent les positions des acides aminés dans la protéine. Une autremutation est une délétion d‘acide aminé (L242_244del) en position 242.
D’après (D’après figure : https://www.calameo.com/accounts/6282760)
- On doit se rendre à l’évidence, à quand le prochain variant du SARS-CoV-2 ? … Un autre variant venu du Brésil apparaît au Japon !
Une mutation, originaire de l’Amazonie brésilienne dont le Japon a rapporté la découverte très récente, est en cours d’analyse et pourrait impacter la réponse immunitaire, selon une déclaration de l’OMS qui évoque « un variant inquiétant » …avec peu de preuves !
Avec ces variants sud-africain et brésilien, certaines données très incomplètes mentionnent que l’immunité ne protégerait pas parfaitement contre ces variants. Aurait-on à faire à des « variants antigéniques » modifiant les capacités antigéniques de la protéine S (l’immunogène) comme évoqué ci-dessus ? Cela, ne semble pas être le cas du variant britannique, que l’on pourrait baptiser de « variant de comportement ».
Cela voudrait dire que l’efficacité des vaccins actuels pourrait être moins bonne. Les vaccins à ARN messager représentent un avantage majeur, ils sont faciles à faire rapidement évoluer, en quelques semaines. Dans une telle situation, pas de panique, il suffira de réaliser une vaccination complémentaire pour le nouveau variant, avec une seule dose de rappel pour des personnes déjà vaccinées. Le vaccin antigrippal évolue bien, pratiquement chaque année ! Il devrait exister de la ressource opérationnelle. Le fondateur et PDG de BioNtech, Uğur Şahin, pense qu’il n’y a aucune raison pour qu’une nouvelle préparation vaccinale ne puisse être créée dans des délais courts : « Technically it is possible to make a new vaccine mimicking the new strain in a few weeks ». La flexibilité de la technologie réside dans le fait que l’information génétique – l’ARN messager – peut être facilement révisée et réécrite et permettre de cibler les dernières formes mutantes du virus. Les autres ingrédients du vaccin (i.e., sels, sucres et nanoparticules lipidiques) n’auraient pas à être modifiés. Selon des responsables de Moderna Therapeutics et du gouvernement américain, les vaccins reprogrammés pour contrer l’émergence de nouvelles souches du virus SARS-CoV-2 pourraient atteindre le marché plus rapidement, sans passer par de grands essais cliniques, selon des responsables de Moderna Therapeutics et du gouvernement américain. La raison : pas besoin de grandes études pour prouver que cette famille de vaccins fonctionne.
- Remarques générales : Antigénicité et efficacité des vaccins
La surveillance génomique des variants du SARS-CoV-2 s’est largement concentrée sur les mutations de la glycoprotéine de pointe (protéine S), qui est impliquée dans la liaison aux cellules ciblées par le virus. C’est aussi une cible majeure des anticorps neutralisants. D’où une réflexion intense pour savoir si les mutations de la glycoprotéine S pourraient limiter l’efficacité des anticorps produits par l’hôte et de ce fait potentiellement compromettre l’efficacité du vaccin. La protéine S est la cible antigénique principale du virus sélectionné pour la préparation des vaccins actuels. Pratiquement, ces vaccins ont été élaborés à partir de gènes, c’est-à-dire d’ARN antérieurs à l’apparition de certaines de ces mutations. Pas de panique, pour le moment (mais c’est un peu tôt pour se prononcer très sérieusement) ça ne semble pas être encore être le cas…En dehors de quelques restrictions sur les variants africains et brésiliens
On peut penser qu’à ce stade de l’épidémie, la forte sélection d’un variant au niveau de la population n’est probablement pas déterminée par les anticorps de l’hôte car il n’y a pas suffisamment d’individus immunisés pour orienter systématiquement le virus dans une direction donnée.
D’autre part, comme les vaccins actuels provoquent une réponse à la protéine S dans son ensemble, on espère qu’une protection efficace pourra encore se produire malgré quelques changements au niveau des sites antigéniques des variants de SARS-CoV-2.
Il est important de séparer la cause de la conséquence pour évaluer correctement les données sur la neutralisation des variants de la protéine S par les anticorps des patients vaccinés.
Quelle que soit la manière dont les mutations ont été sélectionnées (« effet fondateur » ou « sélection naturelle ») au sein de la population virale, il est raisonnable de s’attendre à ce que des mutations de la protéine S puissent affecter la neutralisation du virus par des sérums de convalescents. Pour répondre à de telles questions, il est important de considérer à la fois l’ampleur des modifications des capacités de neutralisation mais aussi le nombre d’échantillons de sérum de convalescents évalués avant de trancher un tel dilemme.
Un autre problème est que les glycoprotéines virales sont sujettes à des changements évolutifs. Ainsi, une mutation qui améliore les propriétés d’un virus, telles que la liaison à son récepteur, pourra en contrepartie réduire une autre de ses propriétés qui serait la capacité d’échapper aux anticorps de l’hôte. En effet, des preuves récentes suggèrent que cela pourrait être le cas pour D614G. Il est possible que les mutations de la protéine S qui sont considérées « utiles » pour le virus en ce moment pourraient l’être moins dans le contexte de l’immunité qui va se développer au niveau de la population à l’avenir.
Définir de telles dynamiques et leur influence potentielle sur l’efficacité vaccinale va certainement nécessiter une surveillance à grande échelle de l’évolution du SARS-CoV-2 mais aussi des capacités immunitaires de l’hôte pendant longtemps. De plus, la préparation des vaccins sur la base des variants détectés doit être prise en compte par anticipation dès que leur découverte avance. Face à l’évolution de la pandémie, il paraîtrait raisonnable d’ouvrir des nouvelles lignes de production des vaccins existants mais aussi de développer de nouveaux vaccins pour contrer les variants et se préparer des revaccinations futures si besoin est[5]. À plus long terme, la vaccination des animaux vecteurs doit également être considérée pour éviter les pertes économiques dans l’agriculture et le débordement de certaines espèces animales à l’homme.
Ces diverses mutations posent des questions sur la notion d’« immunité de groupe ». Selon le paradigme habituel, elle s’installerait naturellement après que le virus a touché un grand nombre d’individus (de 60 à 70 %) et elle empêcherait de ce fait sa circulation. Face à cette pandémie il semble que son atteinte rapide reste assez illusoire. L’immunité de groupe a été surtout décrite qu’après des campagnes de vaccination massives. Des décennies ont parfois été nécessaires pour l’atteindre en ayant des vaccins très efficaces (variole, poliomyélite, tuberculose, rougeole) capables d’induire une immunité sur le long terme. Après des déclarations probablement prématurées, sur l’atteinte d’une « immunité de groupe » à Manaus au Brésil avec plus de 60 % de la population préalablement infectée, il est constaté que les personnes se réinfectent. Seuil insuffisant d’« immunité de groupe » ou apparition d’un variant résistant à cette immunité installée ?
- Nouvelles chaînes de transmission du SARS-CoV-2 – Les débordements interespèces et la survie du SARS-CoV-2
Une publication récente dans Science (8 janvier 2021-vol 371) mérite notre attention. Elle nous rappelle qu’il faut savoir que de nombreuses espèces d’animaux sauvages mais aussi domestiques peuvent aussi véhiculer le SARS-CoV-2 (et des virus apparentés)[6]. Des groupes ont signalé la présence de coronavirus (CoV) dans les chauves-souris « fer à cheval » en Chine.
Et dans les pangolins transférés en contrebande des régions asiatiques du Sud. Aucune des deux espèces ne peut être considérée comme étant progénitrice du SARS-CoV-2 humain pour le moment. Des chats et chiens domestiques, ainsi que des tigres des zoos, se sont également avérés être naturellement infectés par le SARS-CoV-2 par l’homme, mais il n’y a aucune preuve qu’ils peuvent infecter les humains. Il est donc improbable qu’ils soient les hôtes sources du SARS-CoV-2[7].
À ce jour, les infections par le SARS-CoV-2 qui ont été signalées dans les fermes de visons dans huit pays (Pays-Bas, Danemark, Espagne, France, Suède, Italie, États-Unis et Grèce) selon les dernières données de l’Organisation mondiale pour la santé animale ont été suspendues par les sacrifices des animaux. En plus d’une transmission d’animal à humain (et vice versa) dans les fermes élevant des animaux, une autre question est très préoccupante. Elle concerne les chaînes de fournisseurs de produits congelés. Dans diverses villes de Chine, plusieurs petites éclosions de COVID-19 semblent avoir été causées par une contamination virale de fruits de mer non cuits ou de la viande de porc d’importation car il s’est avéré que les signatures du génome viral identifié dans ces flambées étaient différentes de celle des souches chinoises les plus courantes ? Pour des auteurs là est la preuve que le SARS-CoV-2 pourrait survivre jusqu’à 3 semaines dans la viande et sur les surfaces des emballages d’aliments congelés sans une notable perte de ses capacités infectieuses[8] Cette question importante, reste encore très débattue, des stagnations aussi longues du virus dans des produits ou sur des surfaces ont été rarement mentionnées (et peut-être mal recherchées). Le SARS-CoV-2 peut-il être transmis par la prise alimentaire ? Une question à préciser.
Des tentatives d’infections expérimentales et des essais de liaison entre la protéine S de ces virus animaux et son récepteur l’ACE2 a permis de montrer que le SARS-CoV-2 peut avoir de nombreux hôtes. Il est probable que des mutations qui surviennent dans le SARS-CoV-2 chez les animaux seront susceptibles d’augmenter sa pathogenèse ou sa transmissibilité chez les humains. La protéine S à une prédisposition à avoir plus de mutations car elle est la première protéine d’interaction virus hôte et elle fait donc face une plus forte pression de sélection. Compte tenu du risque éventuel des retombées du SARS-CoV-2 entre les humains et certains animaux, il est impératif de suivre de près les mutations dans le génome viral des animaux et des humains, en particulier les régions du génome codant pour la protéine affectant les tests de diagnostic, les cibles des médicaments antiviraux et des vaccins en cours de développement.
- Nouvelles de dernière minute.
La situation évolue de jour en jour et une certaine tension monte à propos de l’approvisionnement en vaccins. Ce 27 janvier 2021, on reparle d’un troisième confinement et les variants obsèdent tout le monde. Tous, experts virologues, politiques, décideurs, espèrent bloquer le plus rapidement possible la transmission de l’épidémie… Mais sont désarmés. Les chercheurs essaient de comprendre pourquoi les souches de SARS-CoV-2 identifiées au Royaume Uni et en Afrique du Sud sont plus infectieuses. Ils s’interrogent afin de savoir si les défenses immunitaires stimulées par les vaccins seront aussi efficaces face à ces souches émergentes[9]. Une prépublication publiée le 7 janvier 2021 rapporte qu’une mutation partagée par les deux variants n’altère pas l’activité des anticorps produits chez les patients vaccinés avec le vaccin Pfizer/BioNTech. Moderna tente aussi de rassurer et a annoncé, lundi 25 janvier, que son vaccin appelé mRNA-1273 permettait bien le développement d’anticorps contre ces deux principaux variants du virus SARS-CoV-2. Au terme d’essais, les experts de la firme assurent dans un communiqué (en anglais) que le vaccin protégera contre les variants détectés jusqu’à la date de cette communication. Il est tout de même mentionné « une réduction par six » des niveaux d’anticorps contre le variant sud-africain (B.1 351). Cependant, malgré cette réduction, les niveaux resteraient au-dessus de ce qui est attendu comme nécessaire pour procurer une protection nous affirme Moderna. Toutefois la firme mentionne que, « par précaution » elle va tout de même mettre à l’essai un vaccin candidat pour un rappel contre le variant sud-africain !
En fait ce sont les résultats sur le terrain qui permettront de trancher. Si les personnes vaccinées sont confrontées au bout de quelques mois à une baisse notable de leur protection sous un niveau critique on peut penser que ces variants pourraient éventuellement provoquer une réinfection. Une telle situation va nécessiter une évaluation du niveau d’anticorps dans le sang ainsi que la définition du profil de leur défense immunitaire par un phénotypage de leurs lymphocytes (i.e., peu probable à grande échelle). Que faire face à une telle évolution défavorable de la pandémie ? Faudra-t-il comme l’envisage Moderna réactiver l’immunité en injectant une troisième dose ? Plus complexe, mais faisable en quelques mois, faudra-t-il changer la séquence génétique de l’ARNm vaccinal afin d’avoir un nouveau vaccin ?
Le vaccin produit par le laboratoire britannique AstraZeneca, conçu en partenariat avec l’université d’Oxford, présente l’avantage d’être moins contraignant à déployer que les deux autres (il peut se conserver dans un simple réfrigérateur et non à très basse température) AstraZeneca fait l’objet de débats en Allemagne en ce qui concerne son efficacité chez les personnes âgées de plus de 65 ans. AstraZeneca dont le vaccin est déjà autorisé et largement déployé au Royaume-Uni, explique dans un communiqué avoir publié une seconde étude, après une première étude réalisée chez des patients ne dépassant pas 55 ans. Les données scientifiques de cette seconde étude, publiées dans la revue The Lancet[10], « démontrent que les personnes âgées ont montré de fortes réponses immunitaires au vaccin, 100 % d’entre elles ayant généré des anticorps spécifiques après la deuxième dose ». Le Laboratoire fait également part de difficultés pour tenir ses engagements à l’égard des livraisons de vaccin ; elles ont irrité Bruxelles. Le feu vert réglementaire de l’UE sera-t-il différé en attendant la clarification d’une situation un peu confuse ?
Pour les laboratoires Français, les mauvaises nouvelles s’accumulent. Le Laboratoire Sanofi, avait déjà annoncé en décembre que son vaccin avait pris un gros retard du fait de résultats moins bons qu’attendu et ne serait disponible que fin 2021. L’Institut Pasteur a annoncé qu’il arrêtait le développement de son principal projet de vaccin contre le SARS-CoV-2, car les premiers essais ont montré qu’il était moins efficace qu’espéré. Son vaccin utilisait comme base une adaptation du vaccin contre la rougeole pour combattre le SARS-CoV-2. Il y a d’autres projets vaccinaux à l’Institut Pasteur, comme un vaccin à usage nasal et un second candidat vaccin à ADN. Ces deux projets sont encore en phase préclinique, aucun essai n’a encore été mené sur des humains… Soyons patients !
On peut se demander, au vu des difficultés de fourniture de certains vaccins et de leurs spécificités, si on ne s’oriente pas vers une guerre des commandes de vaccins contre le coronavirus qui risque de provoquer des dégâts humains. Une guerre dans laquelle les considérations techniques et sanitaires vont se trouver confrontées à des opérations géostratégiques d’envergure. Par exemple, la Chine, via son vaccin CoronaVac de Sinovac, a une influence croissante dans plusieurs régions du globe allant de la Turquie[11], à plusieurs pays du Moyen-Orient et au Brésil. La Russie, l’Inde développent leurs campagnes vaccinales avec des vaccins mal connus du fait d’une communication limitée sur leurs spécificités et leurs capacités de protection.
Quel scénario choisir ? Il y a le scénario optimiste pour certains tenants de la pensée magique selon laquelle, le virus va se fatiguer à l’été et « le vaccin magique », mais difficile à obtenir, va forcément bloquer la transmission de l’épidémie. Tout sera réglé avant la fin 2021. Les itinérances planétaires des hommes d’affaires et des touristes ainsi que l’activité économique vont pouvoir reprendre.
Ne faudrait-il pas tout de même se préparer à un scénario plus pessimiste. Il se pourrait que le virus avec ses divers variants continue à circuler pendant plusieurs années (même si c’est à bas bruit !). Il va falloir apprendre à nous adapter avec un recours à plusieurs types de vaccins et également rechercher de nouvelles armes antivirales qui font largement défaut pour le moment. Comme le faisait récemment remarquer l’immunologiste clinicien, Yves Lévy, « le vaccin est aussi un enjeu de souveraineté ». Doit-on changer de paradigme industriel et repenser la création de médicaments et de vaccins dans notre pays ? Dilemme assez mal garanti par nos industriels et nos dirigeants au cours des vingt dernières années.
Max Lafontan, Directeur de Recherches émérite Inserm,
Secrétaire perpétuel Académie des sciences, Inscriptions et Belles-Lettres de Toulouse.
(Le 16 janvier 2021 modifié le 27 janvier)
[1] L’adaptation apparente du SARS-CoV-2 aux visons est assez préoccupante car l’évolution continue du virus dans un réservoir animal pourrait potentiellement entraîner un passage récurrent d’un nouveau virus SARS-CoV-2 muté du vison à l’homme, voire à d’autres mammifères. Pour cette raison, de nombreux pays ont renforcé la surveillance et, dans certains cas, mis en œuvre des abattages à grande échelle de visons dans les fermes d’élevage.
[2] Il ne faut pas oublier que la saturation des hôpitaux anglais peut déboucher sur un accroissement de la mortalité des patients qui est due à des carences de matériels de réanimation et de soins adaptés. En fait, elle n’aurait rien à voir avec la mutation virale.
[3] Un arbre phylogénétique est un arbre schématisé, ici basé sur des séquences géniques, qui montre les relations de parenté entre des groupes d’êtres vivants (ici SARS-CoV-2).
[4] En biologie systématique, un clade est un groupe de taxons ayant une origine phylétique commune. Le clade contient l’ancêtre commun et la totalité de ses descendants. Tout le monde connait les taxons binomiaux utilisés en biologie pour nommer un animal, une plante..etc. (par exemple, le loup : Canis lupus – le genre Canis (taxon générique) et l’espèce dans le genre lupus (taxon spécifique).
Lire plus d’informations sur la question: https://www.aquaportail.com/definition-1644-clade.html
[5] On notera à l’occasion la position scandaleuse de la Sanofi qui se livre, en cette période critique qui devrait mobiliser tous les acteurs des vaccins, à des délestages de chercheurs sans oublier tout de même de rémunérer grassement ses actionnaires.
[6] Zhou P, Shi ZL. SARS-CoV-2 spillover events. Science. 2021 Jan 8;371(6525):120-122.
[7] B. Hu, H. Guo, P. Zhou, Z. L. Shi, Characteristics of SARS-CoV-2 and COVID-19. Nat. Rev. Microbiol. 10.1038/s41579-020-00459-7 (2020).
[8] Han, X. Zhang, S. He, P. Jia, Can the coronavirus disease be transmitted from food? A review of evidence, risks, policies and knowledge gaps. Environ. Chem. Lett. 10.1007/s10311-020-01101-x (2020)..
[9] Could new COVID-19 variants undermine vaccines? Labs scramble to find out. Nature (2021) 589, 177-178
[10] Voysey M et al. Safety and efficacy of the ChAdOx1 nCoV-19 vaccine (AZD1222) against SARS-CoV-2: an interim analysis of four randomized controlled trials in Brazil, South Africa, and the UK. Lancet 2021; 397: 99–111
[11] Le vaccin inactivé contre le COVID-19 du groupe pharmaceutique chinois Sinovac, le CoronaVac, a montré une efficacité de 91,25 % dans les essais cliniques de phase III conduits en Turquie, a annoncé jeudi 24 décembre le ministère turc de la Santé.
